Détection Directe du poliovirus par Séquencage Nanopore (DDNS) - Selle
Joyce Akello, Alex Shaw, Manasi Majumdar, Catherine Troman, Javier Martin, Nick Grassly, Aine OToole, c.ansley, Erika Bujaki, rachel.colquhoun, khurshida, alammu, Andrew Rambaut, arshady, Shannon Fitz, Ben Bellekom
Abstract
Ce protocole est une mise à jour du protocole décrit dans l'article "Rapid and sensitive direct detection and identification of poliovirus from stool and environmental surveillance samples using nanopore sequencing" par Shaw et al dans le Journal of Clinical Microbiology (2020), DOI : 10.1128/JCM.00920-20 et est généralement connu sous le nom de Direct Detection of Poliovirus by Nanopore Sequencing (DDNS).
Le protocole vise à amplifier la région VP1 du poliovirus par une PCR semi-nichée utilisant une amorce pan-Enterovirus et des amorces spécifiques à la polio, suivie par l'amplification de la région VP1 à l'aide d'un ensemble d'amorces spécifiques pour le polio. Nous utilisons des amorces à code-barres, ce qui simplifie beaucoup le processus ultérieur de préparation de la bibliothèque.
Ce protocole est destiné à être utilisé avec les réactifs de séquençage par ligature chimique du kit 14 d'Oxford Nanopore et peut être utilisé avec le séquenceur MinION Mk1B ou GridION.
Dans les étapes du protocole, des contrôles de qualité sont inclus et suivent le flux de travail défini dans le document "Quality Control and Data Recording for DDNS" (Contrôle de qualité et enregistrement des données pour le DDNS).
English
Title: Direct Detection of poliovirus and Nanopore Sequencing (DDNS) - Stool V.4
Abstract: This protocol is an update from the protocol described in the paper "Rapid and sensitive direct detection and identification of poliovirus from stool and environmental surveillance samples using nanopore sequencing" by Shaw et al in the Journal of Clinical Microbiology (2020), DOI: 10.1128/JCM.00920-20 and is commonly known as Direct Detection of Poliovirus by Nanopore Sequencing (DDNS).
The protocol aims to amplify the VP1 region of poliovirus through a semi-nested PCR using a pan-Enterovirus primer and polio specific primers followed by amplification of the VP1 region using a polio specific primer set. We use barcoded primers as this greatly simplifies the subsequent library preparation process.
This protocol is for use with Oxford Nanopore kit14 chemistry ligation sequencing reagents and can be used with the MinION Mk1B or GridION sequencer.
Within the protocol steps, quality control checks are included and follow the workflow set out in the document "Quality Control and Data Recording for DDNS".
Before start
Ce protocole décrit l'amplification de la région VP1, le codage à barres de l'échantillon et la préparation de la bibliothèque. Nous pensons que les utilisateurs auront effectué une extraction d'ARN avant ce protocole pour extraire l'ARN du poliovirus. Nous recommandons le kit d'isolement de l'ARN viral MagMAX pour ce processus.
Amorces VP1 à code-barres :
Pour simplifier le protocole, nous utilisons une plaque d'amorce à 96 puits avec 5µM d'amorce Y7 à code-barres et 5µM d'amorce Cre à code-barres dans chaque puits, ce qui donne une concentration totale d'amorce de 10µM.
Chaque puits contient des amorces Cre et Y7 avec le même code-barres unique, par exemple A1 = Y7 avec le code-barres 1 et Cre avec le code-barres 1, A2 = Y7 avec le code-barres 2 et Cre avec le code-barres 2, etc.
L'ensemble complet des 96 séquences d'amorces à code-barres figure dans le Dataset_S1 de Shaw et al, 2020 et dans la feuille de calcul ci-jointe. BarcodedPrimers.xlsx
Steps
Sample Organisation
Les paires d'échantillons (avec le même EPID) peuvent avoir des codes-barres consécutifs, mais essayez de ne pas regrouper des échantillons provenant de la même zone géographique. Cela permet de détecter toute contamination croisée potentielle, car il est alors peu probable que des séquences identiques soient détectées dans des échantillons dont les codes-barres sont consécutifs et qui sont adjacents sur la plaque à 96 puits.
Enregistrez les données de l'échantillon et l'ordre des échantillons dans votre fichier CSV. À ce stade, vous pouvez également ajouter toutes les autres métadonnées dont vous disposez pour les échantillons.
Voici un exemple de fichier CSV de code-barres : barcodes.csv
Il est conseillé de modifier le nom du fichier afin qu'il soit unique pour chaque analyse.
Vous devez également inclure les contrôles positifs et négatifs dans votre liste d'échantillons. Un contrôle positif (Coxsackievirus A20 remis en suspension par le NIBSC) et un contrôle négatif (eau) doivent être inclus dans le premier et le dernier lot d'extraction d'ARN de la journée au minimum.
Si des échantillons sont des répétitions d'une série précédente, notez-le dans la colonne "IfRetestOriginalRun".
Si le traitement de l'échantillon a été retardé, par exemple en raison d'un manque de kits d'extraction ou de mises à jour logicielles empêchant l'exécution, notez "Oui" dans la colonne "DelaysInProcessingForDDNS" et indiquez le type de retard dans la colonne "DetailsOfDelays" (détails des retards).
Premier tour PCR (semi-nest)
Préparer un mélange maître en utilisant les volumes de réaction détaillés dans le tableau ci-dessous pour le nombre d'échantillons que vous avez plus les contrôles négatifs. Le mélange réactionnel et l'enzyme SSIII sont fournis dans l'emballage suivant:
Amorce avant: Y7 [GGGTTTGTGTCAGCCTGTAATGA]
Amorces inverses: Cre [TCAATACGGTGTTTGCTCTTGAACTG] (Arita et al. 2015)
nOPV-MM-R [TCGATACGGTGCTTGGATTTAAATTG]
| A | B |
|---|---|
| Réactif | 1 reaction (μL) |
| 2x Mélange réactionnel | 12.5 |
| Mélange SSIII Platinum Taq | 1 |
| Amorces inverses (10μM) | 1 |
| Eau sans nucléase | 4.5 |
Tableau 1 : Contenu du mastermix pour une seule réaction PCR de premier tour. Ce nombre peut être multiplié pour s'adapter au nombre de réactions que vous effectuerez.
Vortexer le mastermix pendant 3 secondes et centrifuger pendant 5 secondes pour rassembler le contenu au fond du tube. Aliquoter 19μL dans chaque tube PCR et ajouter 5μL d'ARN du échantillon ou d'eau sans nucléase pour les contrôles négatifs.
Incuber dans un thermocycleur pendant 30 minutes à 50°C.
Ajouter 1μL de l'amorce avant à chaque réaction.
Amplifier en utilisant ses conditions de cycle suivant| A | B | C | D | | --- | --- | --- | --- | | Cycle | Étapes | Temperature (°C) | Temps | | 1 | Dénaturation initiale | 94 | 2 minutes | | 42 | Dénaturation | 94 | 15 secondes | | | Hybridation des amorces | 55 | 30 secondes | | | Extension | 68 | 2 minutes 30 seconds | | 1 | Prolongation | 68 | 5 minutes | | - | Maintenez | 10 | - |
Tableau 2 : Conditions de cyclage pour la première tour de PCR
Une fois la PCR terminée, vérifiez si des réactions se sont évaporées. Si c'est le cas, notez-le dans l'échantillon CSV.
Seconde tour PCR (VP1 amplification)
L'amplification du VP1 est réalisée à l'aide d'amorces à code-barres comme décrit dans Dataset_S1 dans Shaw et al 2020. Ceux-ci doivent être commandés dans une disposition de plaque à 96 puits et les amorces avant et arrière prémélangées pour obtenir un stock finale de 10μM.
Préparer un mastermix comme décrit ci-dessous en utilisant
| A | B |
|---|---|
| Réactif | 1 Reaction (μL) |
| DreamTaq 2x mastermix | 12.5 |
| Eau sans nucléase | 8.5 |
Tableau 3 : Contenu du mastermix pour la deuxième tour de PCR utilisant des amorces à code-barres.
Vortexer le mastermix pendant 3 secondes et centrifuger pendant 5 secondes pour rassembler le contenu au fond du tube.
Aliquotez 21μL pour chaque puits d'une plaque PCR à 96 puits et ajoutez 2μL d'amorces à code-barres de 10μM (en veillant à utiliser un code-barres différent pour chaque échantillon) et 2μL de produit de PCR de premier tour ou d'eau sans nucléase pour les contrôles négatifs de PCR.
Amplifier en utilisant les conditions de cycle suivantes :
| A | B | C | D |
|---|---|---|---|
| Cycle | Étapes | Température (°C) | Temps |
| 1 | Dénaturation initiale | 95 | 2 minutes |
| 35 | Dénaturation | 95 | 30 secondes |
| Hybridation des amorces | 55 | 30 secondes | |
| Extension | 72 | 1 minute | |
| 1 | Prolongation | 72 | 10 minutes |
| - | Maintenez | 10 | - |
Tableau 4 : Conditions de cyclage pour la PCR VP1
Vérifier tous les contrôles positifs et négatifs de la réaction VP1 sur un gel d'agarose à 1 %. La bande attendue pour le contrôle positif est d'environ 1,2 kb.
Tous les échantillons peuvent être marqués comme "Pass" pour le PositiveControlCheck si tous les contrôles positifs extraits le même jour présentent une bande VP1 sur le gel.
Tous les échantillons peuvent être marqués comme "Pass" pour le NegativeControlCheck si tous les contrôles négatifs extraits le même jour ne présentent pas de bande VP1 sur le gel.
Si l'un des contrôles positifs échoue ou si l'un des contrôles négatifs présente une bande, tous les échantillons doivent être marqués comme échoués.
Si la vérification du contrôle positif échoue, exécuter le(s) produit(s) PCR du premier tour du contrôle positif sur un gel à 1 %.
S'il n'y a pas de bande, répéter la réaction VP1 imbriquée pour le contrôle. Si une bande est visible, éliminer les amplicons VP1 et répéter les réactions VP1 pour tous les échantillons.
Si aucune bande n'est visible après avoir répété la réaction VP1 imbriquée, répéter les extractions d'ARN après avoir vérifié que le kit d'extraction d'ARN est utilisé correctement et qu'il n'est pas périmé.
Si la vérification du contrôle négatif échoue, répéter les deux tours PCR.
Si le contrôle négatif présente toujours une bande sur un gel ou une TapeStation :
Nettoyer soigneusement les stations de travail de PCR et d'extraction d'ARN.
Remplacer tour à tour les réactifs tout en effectuant des réactions à blanc pour déterminer si un réactif a été contaminé.
Effectuer une extraction supplémentaire d'ARN négatif pour confirmer que le kit d'extraction d'ARN n'est pas contaminé.
Préparation de librairies pour ONT MinION : Pooling, End-prep, et ligature d'adaptateurs
Rassembler 2μL de chaque produit PCR VP1 dans un tube de 1,5mL et concentrer avec des billes AMPure.
Ajouter un volume de billes AMPure égal au volume des produits VP1 regroupés et incuber à température ambiante pendant 5 minutes. Secouer doucement les billes après 2 minutes pour faciliter la liaison.
Par exemple, 50 échantillons, 2µl de chaque pool = 100µl du pool, donc ajouter 100ul de billes AMPure.
Centrifuger le tube pendant 3 secondes puis le placer sur un support magnétique jusqu'à ce que toutes les billes ont formé une boulette et que la solution soit claire.
Prélever la solution à l'aide d'une pipette, en évitant de perturber la boulette de billes.
Ajouter 200μL d'éthanol à 80 % dans le tube, laisser reposer pendant 30 secondes, puis retirer et jeter.
Répéter cette étape.
Centrifuger le tube pendant 2 secondes, replacer-le sur l'aimant, puis retirer l'éthanol restant.
Laisser la pastille sécher à l'air libre pendant 30 secondes ou jusqu'à ce qu'elle soit sèche mais ne se fissure pas.
Retirer le tube de l'aimant et ajouter 51μL d'eau sans nucléase. Agiter le tube pour remettre les billes en suspension et incuber à température ambiante pendant 2 minutes.
Centrifuger le tube pendant 3 secondes, puis replacer-le sur l'aimant, pour permettre aux billes de se regrouper complètement.
Prélever 50μL de l'ADN élué et l'ajouter à un tube PCR propre de 0,2mL.
Préparation End-prep :
Ajouter les réactifs suivants de
| A | B |
|---|---|
| Components | Volume (μL) |
| Tampon de réaction UltraII End-prep reaction buffer | 7 |
| Mélange d'enzymes UltraII End-prep enzyme mix | 3 |
Tableau 5 : Réaction pour la préparation finale de votre bibliothèque regroupée
Mélanger doucement en retournant le tube et centrifuger pendant 3 secondes.
Dans le thermocycleur, incuber pendant 5 minutes à 20°C puis 5 minutes à 65°C.
Transférer dans un tube de 1,5 ml et effectuer un nettoyage des billes par AMPure.
Mélanger les billes AMPure à l'aide d'un vortex jusqu'à ce que toutes les billes soient bien mélangées.
Ajouter 60μl de billes remises en suspension dans le tube et agiter le tube pour mélanger.
Incuber à température ambiante pendant 5 minutes. Pousser doucement après 2 minutes pour faciliter la fixation.
Centrifuger le tube pendant 3 secondes puis le placer sur un support magnétique jusqu'à ce que toutes les billes aient formé une boulette et que la solution soit claire.
Prélever la solution à l'aide d'une pipette, en évitant de perturber la boulette de billes.
Ajouter 200μL d'éthanol à 80% dans le tube, laisser reposer pendant 30 secondes, puis retirer et jeter.
Répéter cette étape.
Centrifuger le tube pendant 2 secondes, le replacer sur l'aimant, puis retirer l'éthanol restant.
Laisser la pastille sécher à l'air libre pendant 1 minute ou jusqu'à ce qu'elle soit sèche mais ne se fissure pas.
Retirer le tube de l'aimant et ajouter 61μL d'eau sans nucléase. Pousser le tube pour remettre les billes en suspension et incuber à température ambiante pendant 2 minutes.
Centrifuger le tube pendant 3 secondes, puis le replacer sur l'aimant, pour permettre aux billes de se regrouper complètement.
Prélever 60μL de l'ADN élué et l'ajouter dans un tube propre de 1,5mL.
Du
Centrifuger la NEB Quick T4 Ligase et la placer sur de la glace.
En utilisant le
Centrifuger et décongeler l'adaptateur de ligature (LA) sur de la glace.
Décongeler le tampon de ligature (LNB) à température ambiante, centrifuger , le mélanger par pipetage, puis le placer sur de la glace.
Décongeler le tampon d'élution (EB) et le tampon pour fragments courts (SFB) à température ambiante, mélanger par vortex et placer sur de la glace.
Si vous prévoyez d'utiliser le MinION le même jour, retirez le tampon de rinçage, le flush tether (FLT) et la BSA du congélateur et décongelez-les à température ambiante. Une fois décongelés, placer-les sur de la glace.
Sortez votre FlowCell (FLO-MIN114, R10.4.1) du réfrigérateur pour qu'elle atteigne la température ambiante.
Préparer le mélange suivant en ajoutant les réactifs au tube de 1,5 ml contenant l'ADN préparé :
| A | B |
|---|---|
| Component | Volume (μL) |
| ADN End-prepped | 60 |
| Tampon de ligature (LNB) | 25 |
| Quick T4 Ligase | 10 |
| Adapteur de ligature (LA) | 5 |
Tableau 6 : Mélange réactionnel pour la ligature de l'adaptateur de séquençage
Mélanger doucement en faisant basculer le tube, puis centrifuger le.
Incuber à température ambiante pendant 10 minutes.
Pendant ce temps, vous pouvez effectuer le contrôle du FlowCell
Branchez votre appareil de séquençage (MinION), ouvrez le couvercle et insérez votre FlowCell. Dans le logiciel MinKNOW, naviguez jusqu'au panneau de démarrage et sélectionnez FlowCell check, puis start. Vous saurez alors combien de pores sont disponibles pour le séquençage.
Si votre FlowCell a déjà été utilisée, au lieu d'effectuer un contrôle du FlowCell, lancez un séquençage fictif en sélectionnant Start Sequencing, nommez le séquençage "flowcell_check", sélectionnez n'importe quel kit, puis réglez la durée pour 10 minutes, passez à l'examen final et lancez le séquençage. Au début de l'exécution, une courte vérification du FlowCell sera effectuée et un nombre plus précis de pores disponibles sera donné (calculé en additionnant les pores disponibles et indisponibles dans le graphique d'état des pores (voir l'image d'exemple ci-dessous)).
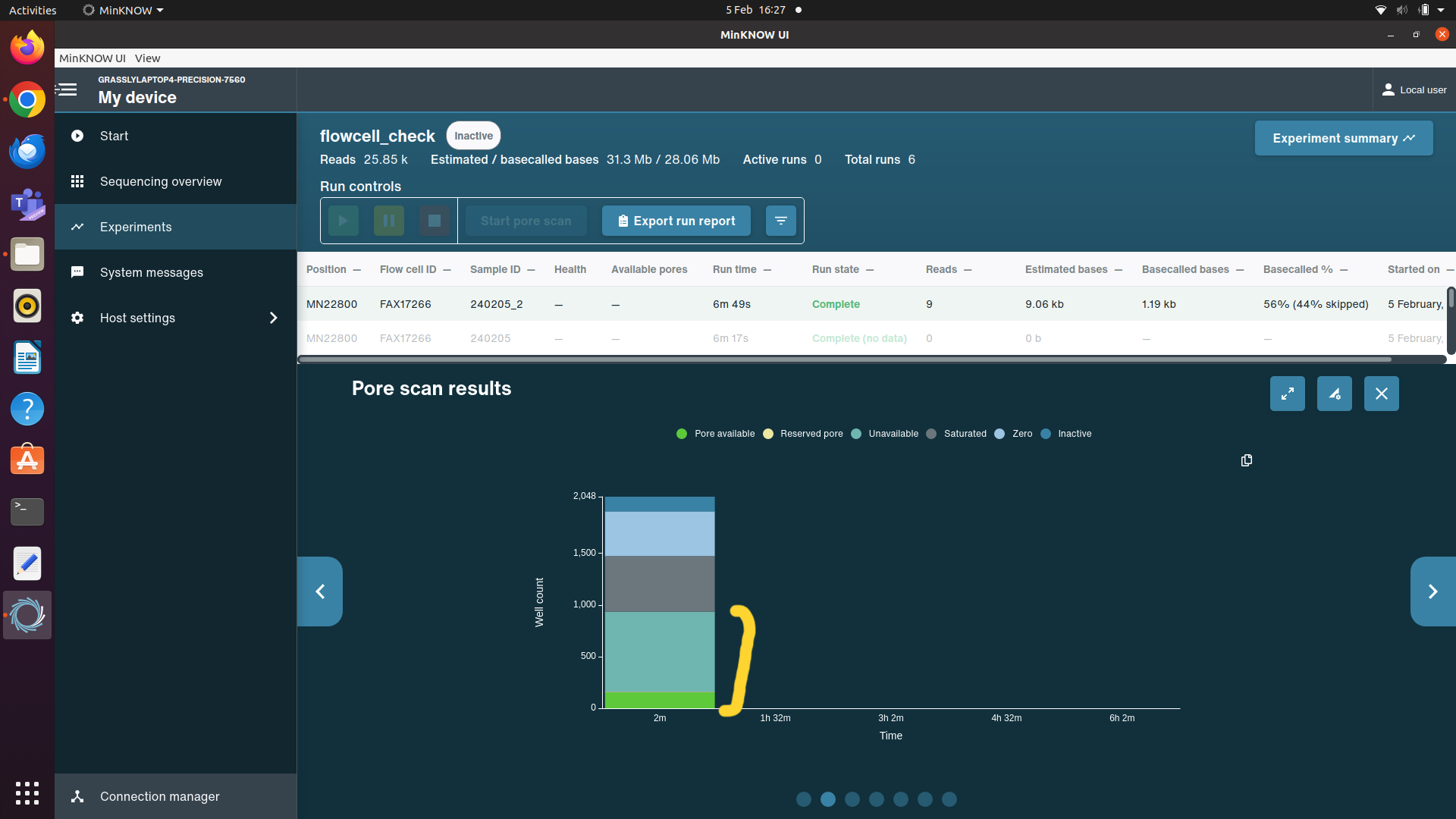
Si un FlowCell a moins de 700 pores, ne l'utilisez pas pour une analyse DDNS de 96 échantillons. Prenez une autre et effectuez une autre vérification. Le nombre de pores disponibles dans le FlowCell utilisée doit être noté dans l'échantillon CSV dans la colonne "PoresAvilableAtFlowCellCheck" et l'ID du FlowCell doit être enregistré dans la colonne "FlowCellID". Notez également le nombre de fois où le FlowCell a été utilisée dans la colonne "FlowCellUses".
Effectuer une purification des billes AMPure en utilisant 40μL de billes AMPure XP remises en suspension.
Mélanger les billes AMPure à l'aide d'un vortex jusqu'à ce que toutes les billes soient bien mélangées.
Ajouter 40μL de billes remises en suspension dans le tube de 1,5mL et mélanger en faisant basculer le tube.
Incuber à température ambiante pendant 5 minutes. Pousser doucement après 2 minutes pour faciliter la fixation.
Centrifuger le tube pendant 3 secondes puis placer-le sur un support magnétique jusqu'à ce que toutes les billes ont formé une boulette et que la solution soit claire.
Prélever la solution à l'aide d'une pipette, en évitant de perturber la boulette de billes.
Ajouter 250μL de tampon pour fragments courts (SFB). Retirer le tube de l'aimant et remettre les billes en suspension dans le SFB en faisant basculer le tube.
Centrifuger le tube pendant 3 secondes, puis le replacer dans l'aimant.
Laisser les billes se regrouper, puis les retirer et les jeter.
Répéter cette étape.
Centrifuger le tube pendant 2 secondes, le replacer sur l'aimant, puis retirer toute trace de SFB.
Laisser la boulette sécher à l'air libre pendant 1 minute ou jusqu'à ce qu'elle soit sèche mais non fissurée.
Retirer le tube de l'aimant et ajouter 15μL de tampon d'élution (EB). Pousser doucement pour remettre en suspension les billes et incuber à température ambiante pendant 10 minutes.
Centrifuger le tube pendant 3 secondes, puis le replacer sur l'aimant, pour permettre aux billes de se regrouper complètement.
Prélever 12µl de l'ADN élué et le transférer dans un tube propre de 1,5ml.
Amorçage et chargement de la Flowcell MinION
Décongeler le tampon de séquençage (SB), les billes de librairie (LIB), le Flow Cell Tether (FCT) et un tube de Flow Cell Flush (FCF) à température ambiante, puis les placer sur de la glace.
Vortexer le SQB, le FCF et le FCT, centrifuger et remettre sur la glace. Centrifuger la LIB et la remettre sur la glace.
Pour créer l'amorçage, mélanger les réactifs suivants dans un tube propre de 1,5 ml :
| A | B |
|---|---|
| Réactif | Volume (μL) |
| Flow cell flush (FCF) | 1,170 |
| Flow cell tether (FCT) | 30 |
| BSA (50mg/ml) | 5 |
Mélanger par pipetage et centrifuger . Placer sur de la glace jusqu'au moment de l'utilisation.
Ouvrez le couvercle du dispositif de séquençage par nanopores et faites glisser le couvercle du port d'amorçage du FlowCell dans le sens des aiguilles d'une montre pour que le port d'amorçage soit visible. Après avoir ouvert le port d'amorçage, vérifier qu'il n'y a pas de bulles sous le couvercle. Aspirer un petit volume pour éliminer les bulles (quelques µl). Vérifier visuellement qu'il y a un tampon continu à partir de l'orifice d'amorçage à travers le réseau de capteurs.
À l'aide d'une pipette P1000, chargez lentement 800μL du mélange d'amorçage dans le FlowCell via le port d'amorçage.
Laisser une petite quantité de liquide à l'extrémité de l'embout de la pipette pour éviter d'introduire de l'air dans la FlowCell.
Laisser reposer pendant 5 minutes.
Mélanger le contenu du tube LIB à l'aide d'une pipette juste avant de l'ajouter au mélange de librairies suivant dans un tube de 1,5 ml :
| A | B |
|---|---|
| Réactif | Volume (μL) |
| Bibliothèque d'ADN | 12 |
| Tampon de séquençage (SB) | 37.5 |
| Billes de librairie (LIB) | 25.5 |
Compléter l'amorçage de le FlowCell en ouvrant le couvercle du port SpotOn et en chargeant soigneusement 200μL du mélange d'amorçage dans le port d'amorçage . Comme précédemment, laissez une petite quantité de liquide au fond de l'embout pour éviter l'introduction de bulles d'air.
Lors de l'ajout du mélange d'amorçage, il se peut qu'une petite quantité de liquide remonte par l'orifice SpotOn. Si c'est le cas, faites une pause et laissez le liquide retourner dans le FlowCell avant de continuer à ajouter le mélange d'amorçage.
Mélanger doucement le mélange de librairies préparé en pipettant.
Ajouter le mélange de librairies aux FlowCell via le port SpotOn, goutte à goutte, en laissant chaque goutte s'écouler avant d'ajouter la suivante.
Replacez le couvercle du port SpotOn et fermez le port d'amorçage, puis replacez le couvercle de votre appareil de séquençage.
Ouvrez le logiciel ONT MinKNOW et suivez les étapes ci-dessous pour configurer et démarrer votre cycle de séquençage.
Cliquez sur start, puis sur start sequencing.
Créez un nom pour votre série de séquençage. Il est conseillé de rendre ce nom unique et identifiable si vous avez besoin de revoir les données. Il est recommandé d'indiquer la date et le nom de l'expérience. Dans le nom de l'échantillon, vous pouvez mettre un numéro ou répéter le nom de l'expérience - ce n'est pas aussi important que le nom de la série. Cliquez ensuite sur continuer.
Sélectionnez le kit utilisé - il s'agit de SQK-LSK114. Une fois que vous avez cliqué sur ce bouton, les options de codage à barres apparaissent. Sélectionnez EXP-PBC096, puis cliquez sur continuer.
Dans les options de durée d'exécution, réglez la durée d'exécution sur 4 heures pour une exécution DDNS prospective. Cliquez sur continuer.
Dans les options d'appel de base, sélectionnez l'appel de base de haute précision. Dans les options de codage à barres, assurez-vous que le codage à barres est activé et basculez pour utiliser le code à barres aux deux extrémités. Cliquez sur continuer jusqu'à ce que vous atteigniez l'aperçu de l'exécution, où vous pouvez vérifier les options sélectionnées, puis cliquez sur démarrer l'exécution.
Dans votre échantillon csv, enregistrez le numéro d'exécution dans la colonne "RunNumber", la date dans "DateSeqRunLoaded" et la durée de l'exécution dans "RunHoursDuration".
Lavage du FlowCell
Une fois le séquençage terminé, vous pouvez laver votre FlowCell pour éliminer la librairie restante et préparer un autre séquençage ou pour stocker à 4°C . Le lavage utilise les réactifs fournis dans le kit de lavage ONT :
Décongeler le diluant de lavage à température ambiante et mélanger brièvement au vortex.
Centrifuger le tube de mélange de lavage (WMX) et le placer sur de la glace.
Préparer la solution de lavage suivante dans un tube propre de 1,5 ml
| A | B |
|---|---|
| Component | Volume (μL) |
| Diluant de lavage | 398 |
| Mélange de lavage | 2 |
Ouvrez l'orifice d'amorçage et, à l'aide d'une pipette P1000, prélevez avec précaution une petite quantité de liquide pour éliminer toute bulle d'air sous l'orifice.
Ajouter avec précaution 200μL de la solution de lavage par l'orifice d'amorçage, en laissant une petite quantité de liquide dans l'embout vers la fin pour éviter d'introduire une bulle d'air. Fermer le port d'amorçage et incuber pendant 5 minutes à température ambiante.
Ajouter les 200μL restants de solution de lavage par l'orifice d'amorçage. Fermer l'orifice d'amorçage et incuber à température ambiante pendant une heure.
À ce stade, vous pouvez retirer tous les déchets du canal d'évacuation, en veillant à ce que l'orifice d'amorçage soit fermé avant de le faire.
Vous pouvez également coller une étiquette sur l'emballage pour indiquer la date à laquelle elle a été utilisée, ce qu'elle a subi et pendant combien de temps, par exemple : 13/03/2024 DDNS-run30 4 heures.
Si vous souhaitez conserver la Flow Cell pour une réutilisation ultérieure, retirez le flacon de tampon de stockage du kit de lavage pour le décongeler à température ambiante.
Après l'incubation, vous pouvez soit suivre les étapes d'amorçage et de chargement de la Flow Cell à partir de l'étape 22 pour charger une nouvelle série, soit les étapes suivantes pour stocker la Flow Cell en vue d'une utilisation ultérieure.
Vortexer brièvement le tampon de stockage décongelé pour le mélanger, puis ajouter 500μL lentement par le port d'amorçage.
Fermer l'orifice d'amorçage avant de retirer tous les déchets du canal d'évacuation. Replacer la FlowCell dans sa boîte en plastique et son enveloppe, puis la conserver à 4 °C.

