Загрузка сиквенсов в базу данных коллекции (Specify 7) и экспорт данных в GenBank и GBIF
Nina Filippova, Elena Zvyagina
Abstract
Protocol for uploading sequences and their metadata into the Specify 7 database and subsequent export of data to GenBank and GBIF.
- The first stage of preparing sequences involves their initial processing (trimming ends, assembling), saving the edited sequence, and adding comments to the laboratory journal.
- The second stage involves the actual uploading of sequences, their metadata, and trace files into the Specify 7 database. The database serves as the primary long-term storage location for the sequences. From here, the data is exported to GenBank and GBIF.
- Exporting data to GenBank is done by creating a request in Specify and then uploading the table and sequences to GenBank through batch upload.
- Exporting sequence data to GBIF is done for all sequences open for publication in the database after receiving the corresponding numbers in GenBank. The export of sequences is done through a query template, exporting to a .csv table, and then importing the table into the Integrated Publishing Toolkit portal, where each sequence is linked to the corresponding specimen in the collection dataset.
Before start
Для выполнения протокола необходимо:
-
Установить программное обеспечение для анализа хроматограмм,
-
Зарегистировать аккаунт и получить доступ к соответствующим инструментам в ИС "Specify 7" (http://specify.ugrasu.ru/),
-
Зарегистрировать аккаунт на портале GenBank (https://www.ncbi.nlm.nih.gov/),
-
Зарегистрировать аккаунт и получить доступ к соответствующим инструментам в IPT (http://ipt.ugrasu.ru:8080/),
-
Получить доступ к лабораторному журналу и хранилищу сиквенсов лаборатории.
Steps
Подготовка сиквенсов
Первая стадия подготовки сиквенсов включает их первичную обработку (обрезание концов, ассемблинг, и другие операции по необходимости), сохранение готовой отредактированной последовательности и комментарии в лабораторном журнале.
Создать проект для первичного редактирования сиквенсов в Codon Code Aligner или другом программном обеспечении. Загрузить исходные последовательности с исходными именами (номерами пробирок).
Провести анализ и необходимое редактирование сиквенсов:
- Если сиквенс слишком короткий, грязный или не отвечает другим параметрам качества, напротив этого сиквенса в лабораторном журнале (поле SeqQualRemarks) ставится пометка "плохой" и в дополнительном поле комментарий (поле REMARKS) - рекомендация (сделать с нуля, поставить вторично секвенсовую, поставить обратную секвенсовую и др). Такой сиквенс не загружается в Specify.
- Если сиквенс не достаточной длины, но хорошего качества, в лабораторном журнале (поле SeqQualRemarks) ставится пометка "хороший" и в дополнительном поле комментария (поле REMARKS) - "сделать обратный". Такой сиквенс редактируется и загружается в Specify, оставляя поле сиквенса пустым (оно будет заполнено после получения обратной последовательности).
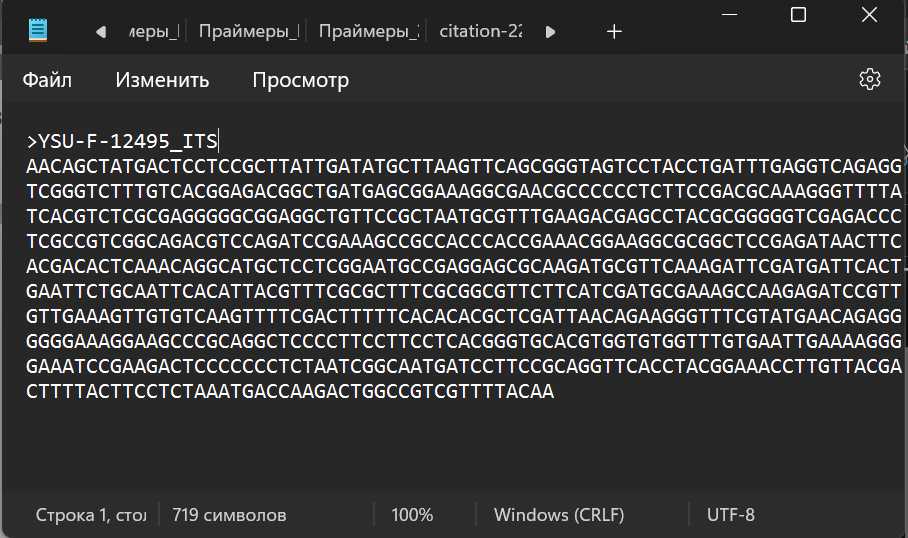
- Если сиквенс достаточно хороший, его редактируют, проверяют по базе NCBI утилитой BLAST и сохраняют в формате FASTA, в первой строке .fas файла после знака > вносят номер образца с акронимом гербария и через нижнее подчеркивание указывают локус .
-
Результат проверки по базе NCBI вносят в соответствующие поля в лабораторном журнале (поля BLAST_closest_species, genSequence FASTA, BLAST_closest_ID, TYPE_specimen), а затем загружается в Specify в полном объеме (см. ниже).
| A | B | C | D | E | F | G | H | I | J | K | L |
|---|---|---|---|---|---|---|---|---|---|---|---|
| № пробирки | № в коллекции | Вид в коллекции | BLAST ближайшее сходство | Имя исходного файла | Последовательность | % BLAST | BLAST closest ID | TYPE specimen | GenBankID | Seq_Qual_Remarks | Work_remarks |
| 173 | YSU-F-12619 | Lentinus castoreus | Lentinellus sublineolatus | 173_ITS_F_A09_01_2022-10-06-23-24-52.ab1 | seq | 99.4 | NR_119505.1 | TENN:059307 | OQ450397 | хороший | короткий, сделать обратный |
| 173 | YSU-F-12619 | Lentinus castoreus | Lentinellus sublineolatus | 173_ITS_R_A08_01_2023-02-15-16-27-33.ab1 | хороший | ||||||
| 174 | YSU-F-12628 | Hypsizygus ulmarius | Hypsizygus marmoreus | 174_ITS_F_B09_02_2022-10-06-23-24-52.ab1 | seq | 99.0 | OP980886.1 | OQ450398 | хороший | короткий, сделать обратный | |
| 174 | YSU-F-12628 | Hypsizygus ulmarius | Hypsizygus marmoreus | 174_ITS_R_B08_02_2023-02-15-16-27-33.ab1 | хороший | ||||||
| 175 | YSU-F-12594 | Cortinarius adustoremosus | Cortinarius adustorimosus | 175_ITS_F_C09_03_2022-10-06-23-24-52.ab1 | seq | 99.0 | NR_172319.1 | PC:R. Henry 883 | OQ450391 | хороший | короткий, сделать обратный |
| 175 | YSU-F-12594 | Cortinarius adustoremosus | Cortinarius adustorimosus | 175_ITS_R_A07_01_2023-02-14-17-16-01.ab1 | хороший |
Пример фрагмента таблицы лабораторного журнала с анализом качества полученных последовательностей и оценкой ближайшего сходства
Загрузка сиквенсов в БД Specify 7
Вторая стадия - собственно загрузка сиквенсов, их метаданных и исходных файлов в БД Specify 7. База данных является основным местом хранения сиквенсов на долгую перспективу. Отсюда данные экспортируются в GenBank и GBIF. Внесение данных на этом этапе требует внимательного отношения.
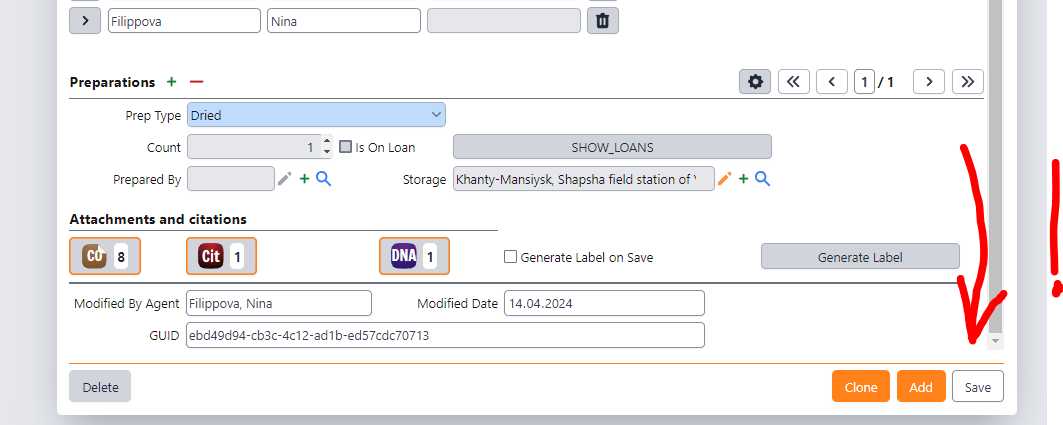
Войти в БД Specify 7 под своим логином. Сделать поиск и открыть карточку соответствующего образца коллекции.
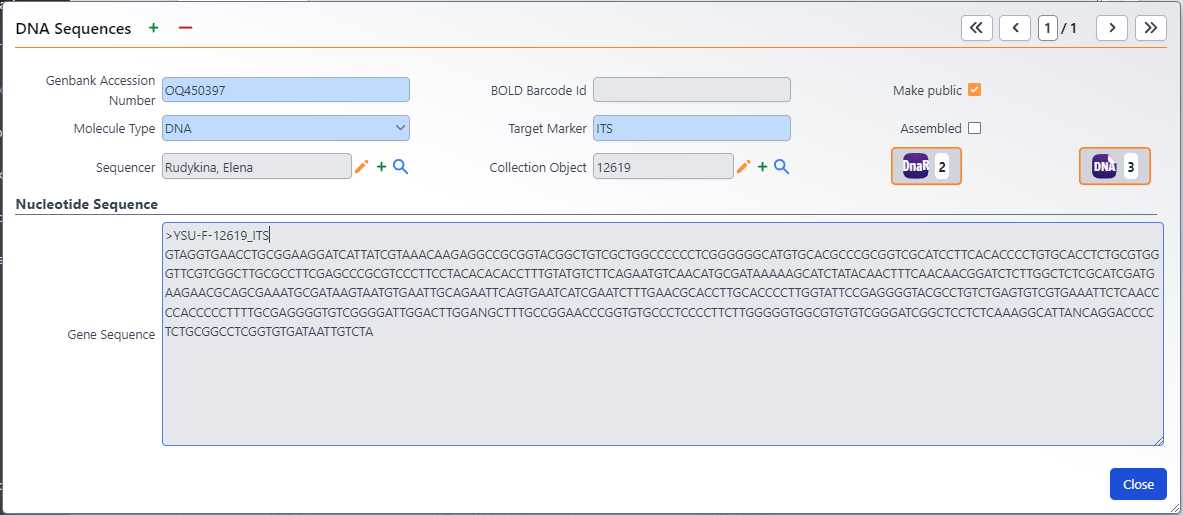
В нижней части формы, открыть кнопку DNA sequence. Внести данные в поля:
- Genbank Accession Number - обязательное поле. Если номера еще нет, внести название фаста-файла без знака > (например, "YSU-F-12619_LOCUS")
- Molecule type - по умолчанию "DNA" (или выбрать иное)
- Sequencer - выбрать ФИО ответственного за секвенирование
- BOLD Barcode ID - добавить уникальный номер BOLD, если имеется
- Target Marker - по умолчанию "ITS", можно выбрать соответствующий локус
- Collection object - заполняется автоматически при сохранении
- Make public - поставить галочку, если сиквенс открыт для публикации в GenBank и GBIF, открывается после получения номера генбанка
- Assembled - поставить галочку, если получена консенсусная последовательность из прямого и обратного (или других) ридов
- Nucleotide Sequence - вставить последовательность в формате FASTA. См. пример ниже
- Отредактировать последовательность так, чтобы в названии последовательности был номер коллекции в формате ">YSU-F-12619_LOCUS", без кавычек, вместо LOCUS соответствующий маркер ДНК
- Открыть следующую форму: DnaR (DNA Sequencing Runs), в которой будет внесена информация об исходных файлах.
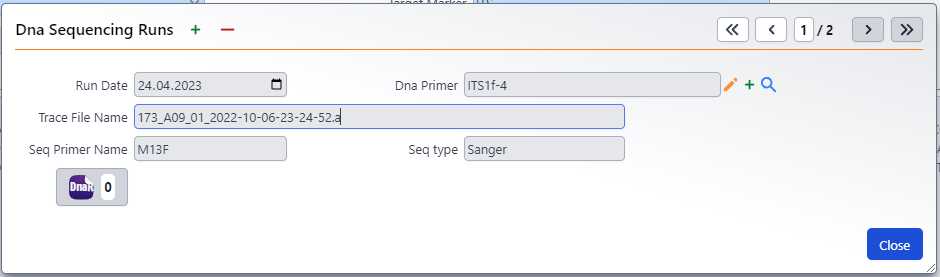
Открыть кнопку DnaR (DNA sequence runs) . Внести данные в поля:
-
Trace File Name - название .ab1 файла исходного сиквенса
-
Run Date - скопировать дату из названия .ab1
-
Seq Primer Name - название праймера секвенсовой реакции (в нашем случае при использовании адаптеров используется 2 значения: M13F, M13R). При использовании для ПЦР праймеров без адаптеров, секвенирование производится с праймерами для ПЦР. Соответственно в этом случае указывается соответствующий праймер
-
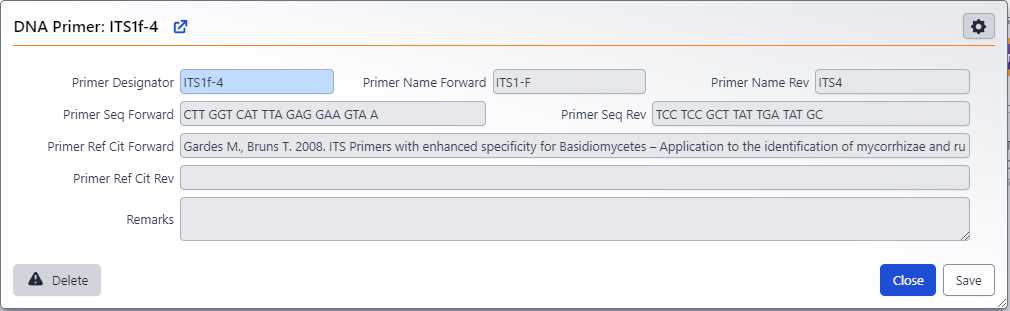
DNA Primer - название пары праймеров, использованных во время ПЦР (выбор из выпадающего списка)
5. Seq Type - тип секвенирования (по умолчанию "Sanger")NoteВ базу данные внесено большое число используемых праймеров и их метаданные. Ввод новых пар понадобится в исключательных случаях использования новых праймеров. В этом случае нужно нажать значок "+" рядом с полем DNA Primer и внести данные в открывшуюся форму -
Если исходных праймеров несколько (например, прямой и обратный), то заполнить следующий файл, нажав "+" в левом верхнем углу формы
-
После заполнения всех форм, нажать "закрыть".
Прикрепить исходные .ab1 файлы сиквенсов :
- Прикрепить исходные файлы сиквенсов можно в форме DNA Seq Runs или DNA Sequences
- Мы пользуемся DNA Sequence Attachments
- Открыть форму, нажать "+", перетащить соответствующий файл, и т.д. (метаданные файла заполняются автоматически)
- По завершении нажать "Close" формы DNA Sequences
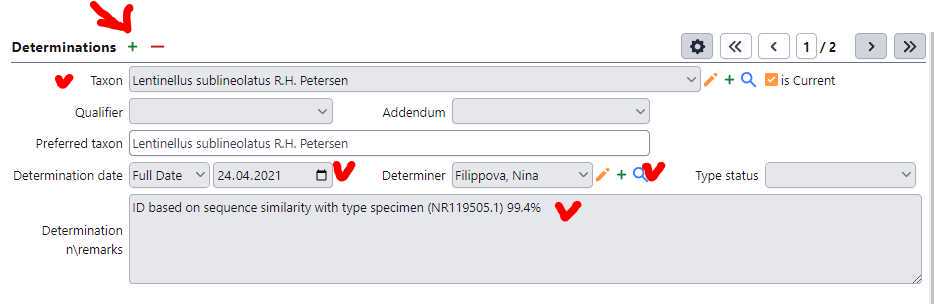
Добавить новую детерминацию таксона на основе поиска ближайших последовательностей в NCBI:
- Добавить новую детерминацию к уже существующей истории определений (нажать "+" рядом с заголовком формы Determinations)
- Добавить новый таксон
- Добавить ФИО того, кто выполнил поиск в BLAST
- Добавить дату (когда выполнен поиск, или когда был получен сиквенс)
- В поле "Remarks" добавить комментарий по шаблону:
- Closest type specimen sequence (NR_119505) 99.4%
- Closest voucher specimen sequence (TD119503) 99.1%
Экспорт данных в GenBank
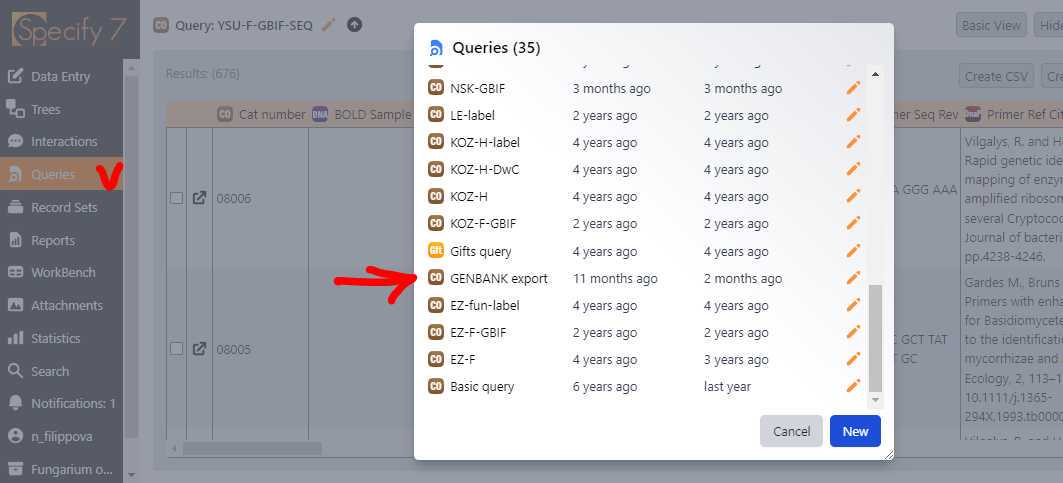
Экспорт данных в GenBank осуществляется через создание запроса в Specify и последующую загрузку таблицы и сиквенсов пакетным образов в GenBank.
В открывшихся настройках запроса в поле Cat Number с включенным оператором "In" ввести необходимые образцы для экспорта в GenBank (например: 12619, 12628, 12594).
Экспортировать созданный запрос в виде таблицы .csv.
Подготовьте файл с метаданными сиквенса (модификаторами). Для этого скачайте шаблон таблицы формата .tsv с портала GenBank или создайте файл с соответствующими названиями полей:
| A | B |
|---|---|
| Sequence_ID | Идентификатор сиквенса, должен совпадать с названием сиквенса в фаста файле (YSU-F-12594) |
| Organism | Название таксона на латыни (Cortinarius adustorimosus) |
| Specimen-Voucher | Номер ваучерного образца (YSU-F-12594) |
| Country | Страна (Russia) |
| Latitude-Longitude | Широта и долгота в формате (61.04119 N 69.31318 E) |
| Collection-Date | Дата сбора образца (03.10.2023) |
| Collected-By | Автор находки (Bulyonkova, Tatiana) |
| Fwd-Primer-Name | Название прямого праймера (ITS1-F) |
| Rev-Primer-Name | Название обратного праймера (ITS4) |
| Fwd-Primer-Seq | Последовательность прямого праймера |
| Rev-Primer-Seq | Последовательность обратного праймера |
Пример заполнения полей модификаторов (метаданных сиквенса) при загрузке последовательностей в GenBank пакетным образом
Перенесите данные из скачанной из Specify таблицы в шаблон. Отредактируйте формат данных в поле географических координат, Страна и Дата. Сохраните шаблон с данными в виде .tsv файла.
Подготовьте файл с сиквенсами в формате Fasta: скопируйте последовательности из столбца экспортированной таблицы Gene Sequence в текстовый файл. Убедитесь, что между названием сиквенса и его последовательностью есть перенос строки или добавьте переносы. Сохраните файл с расширением .fasta или .txt.
Зайдите на портал NCBI (https://www.ncbi.nlm.nih.gov/) под своим логином.
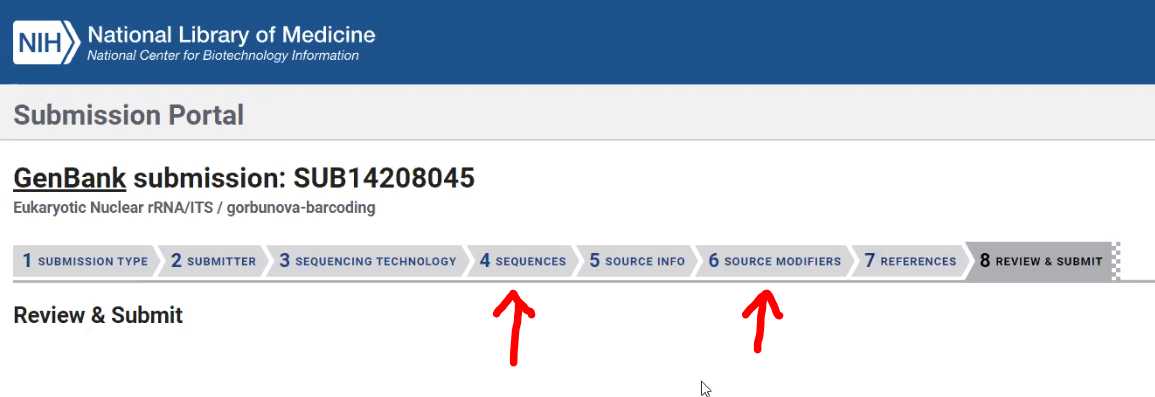
Пройдите последовательно шаги ввода данных для отправки сиквенса:
- Submit
- GenBank
- New Submission
- Submission Type (ribosomal RNA (rRNA) or rRNA-ITS; Eukaryotic nuclear rRNA or rRNA-ITS; contains rRNA-ITS region)
- Submitter (Group 1 - настройте группу соавторов для каждого случая пакетного импорта)
- Sequencing technology (Sanger dideoxy sequencing)
- Assembly state (Unassembled sequence reads или Assembled sequences)
- Sequences (на этом шаге загружается подготовленный файл с сиквенсами в формате fasta. В случае, если появляются предупреждения об ошибках, файл можно отредактировать и загрузить еще раз).
- Source info (Specimen - Voucher)
- Source midifiers (на этом шаге выбираем "upload a tab-delimited table" и загружаем подготовленный файл с модификаторами)
- References (выбираем авторов сиквенсов, публикацию/проект и ее авторов из ранее созданных групп, или создаем заново)
- Revew and Submit (проверяем результат загрузки и нажимаем Submit ).
После получения уникальных номеров сиквенсов в GenBank, зайдите в базу данных Specify и добавьте номера в соответствующее поле в шаблоне DNA Sequence.
Экспорт данных сиквенсов в GBIF
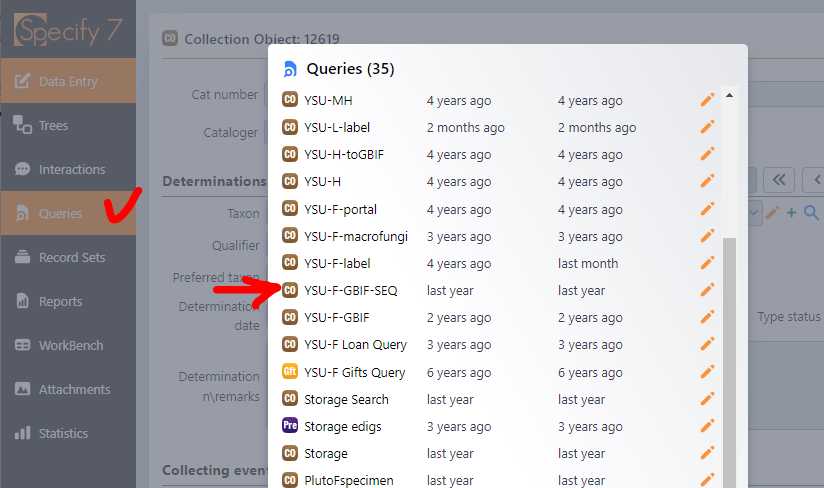
Экспорт данных сиквенсов в GBIF осуществляется для всех открытых для публикации сиквенсов в базе данных после получения соответствующих номеров в GenBank. Публикация осуществляется регулярно раз в месяц (в первых числах каждого месяца). Экспорт сиквенсов осуществляется через шаблон запроса, экспорт в таблицу .csv, и последующий импорт таблицы на портал Integrated Publishing Toolkit, где каждый сиквенс получает привязку к соответствующему образцу набора данных коллекции.
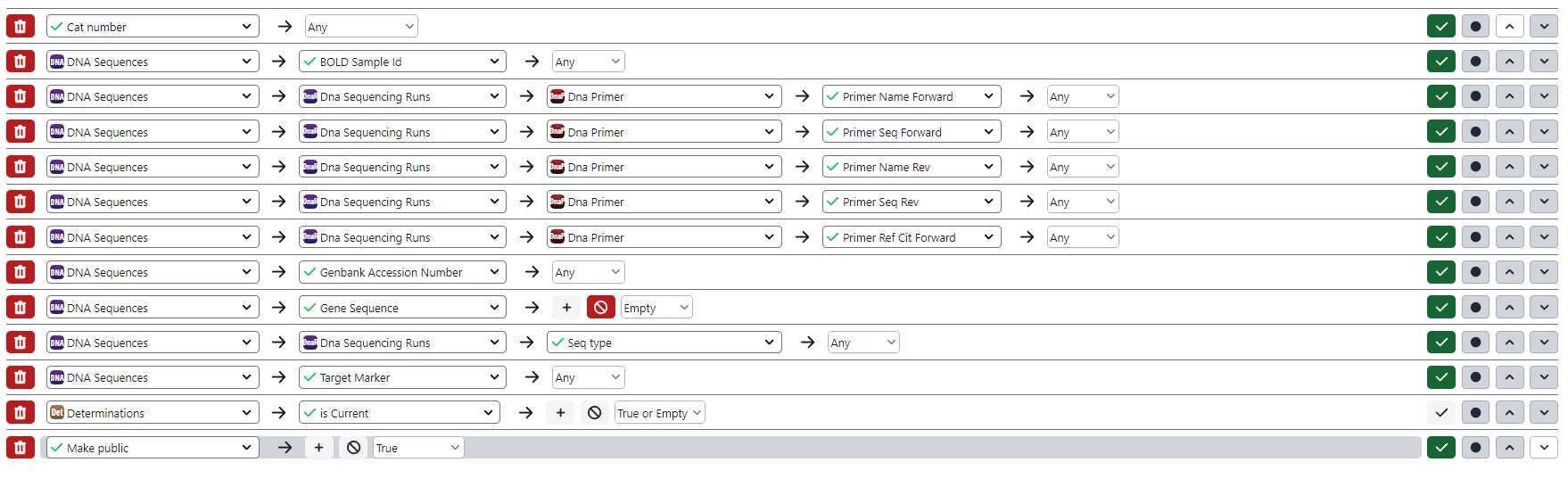
В открывшейся форме запроса можно настроить редактирование на экспорт по заданным параметрам, или использовать поиск всех имеющихся последовательностей:
- CatNumber - номер образца в коллекции (используя оператор "In" можно перечислить интересующие номера через запятую)
- BoldSampleID - номер сиквенса в BOLD
- Primer Name Forward - название прямого праймера (ПЦР)
- Primer Name Reverse - название обратного праймера (ПЦР)
- Primer Seq Forward - последовательность прямого праймера
- Primer Seq Reverse - последовательность обратного праймера
- Genbank Accession Number - номер в генбанке
- Gene Sequence - используя оператор "Not Empty" настроен экспорт только заполненного поля с последовательностью сиквенса. В противном случае поиск выдаст все 10K образцов в базе данных
- Seq type - тип секвенирования
- Target Marker - маркер
- Make Public - True (запрос только тех сиквенсов которые разрешены для публичной публикации)
- Determinations | is Current - поле "текущая детерминация" с оператором "True or Empty" помогает отфильтровать только последние детерерминации, если их было несколько у одного образца. В противном случае поиск выдаст примерно в 2 раза больше записей, номера образцов при этом будут дублироваться.
Загруженная таблица может потребовать доработки: добавления нулей в поле номеров коллекции (5-значный номер), или другие погрешности при вводе данных (лучше редактировать сразу в базе данных и повторить экспорт).
Загрузка таблицы сиквенсов на портал IPT и публикация в GBIF
Публикация данных в GBIF происходит через размещение исходных данных на портале Integrated Publishing Toolkit. В случае загрузки сиквенсов, используется специальное расширение DNA-derived-data, которое позволяет связать таблицу сиквенсов с таблицей загруженных ранее образцов коллекции.
Зайти на портал IPT под своим логином (http://ipt.ugrasu.ru:8080/). Открыть интересующий набор данных на портале IPT (в нашем случае The Fungarium of Yugra State University), начать редактирование набора данных.
В разделе Darwin Core Mappings добавить новое картирование и связать поля таблицы сиквенсов с полями IPT. Отредактировать и сохранить картирование и перейти к редактированию набора данных.
! связывание полей пройдет автоматически, если в исходной таблице названия столбцов переименовать в названия полей в IPT.
| A | B |
|---|---|
| Название поля в Specify | Название для импорта в IPT |
| Cat number | occurrenceID |
| Primer Name Forward | pcr_primer_forward |
| Primer Seq Forward | pcr_primer_reverse |
| Primer Name Rev | pcr_primer_name_forward |
| Primer Seq Rev | pcr_primer_name_reverse |
| Primer Ref Cit Forward | pcr_primer_reference |
| Genbank Accession Number | url |
| Gene Sequence | DNA_sequence |
| Seq type | seq_meth |
| Target Marker | target_gene |
Шаблон для переименования заголовков столбцов таблицы сиквенсов для импорта в IPT
Обновить метаданные набора данных: если идет обновление только сиквенсов, то обновить информацию об общем числе полученных сиквенсов и прошедших баркодинг образцов в Абстракте и Методике работы. Нажать Publish и подождать некоторое время, пока пройдет обновление набора данных в GBIF.
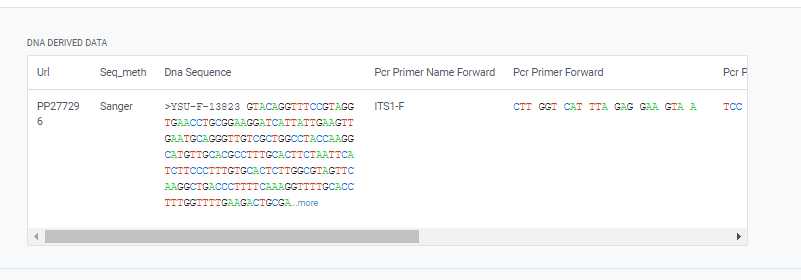
Проверить несколько загруженных образцов в наборе данных в GBIF. Таблица с сиквенсами появится в нижней части карточки образца в разделе Dna Derived Data.
Источники информации о формате данных DNA-Derived-Data и публикации данных последовательностей в GBIF: