在生命科学的微观世界里,单细胞测序技术让我们得以倾听每一个细胞的“低语”。 然而,传统的短读长测序就像“盲人摸象”, 通过零碎的片段推测整体,只能得到基因表达的轮廓,无法描绘出转录本异构体千变万化的全貌。而这些被忽略的异构体信息,却往往包含理解细胞类型、状态和功能差异的关键。现在,PacBio Kinnex 单细胞全长转录组解决方案带来了革命性的突破,它使单细胞测序不再局限于“摸到象腿或象鼻”,以HiFi长读长、高精度和高通量的优势,直接对单个细胞中全长RNA分子进行完整测序,涵盖所有曾经被短读长“错过”的转录异构体细节,同时精准捕获细胞Barcode和UMI信息,使研究人员真正告别“盲人摸象”的时代,能够站在单个细胞的维度看清哪些特定异构体被表达,从而解读出每个细胞内部更丰富、更精确的“完整故事”,为癌症肿瘤免疫和神经科学等领域带来前所未有的洞察。
01 Kinnex single-cell RNA产品优势
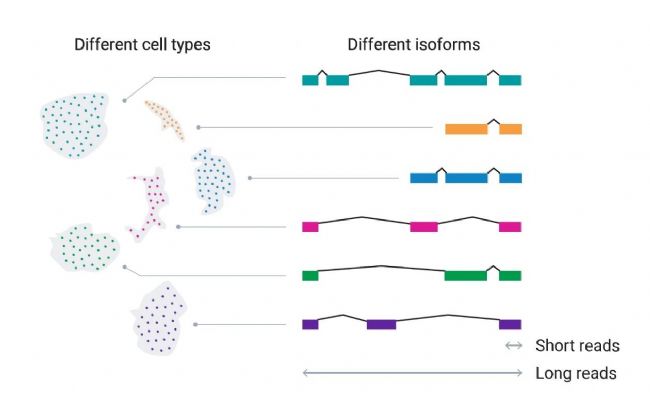
长读长
长读长可直接获得从5'至3'端的全长转录本信息,便于检测到新基因和转录异构体。
高准确性
HiFi测序的准确率可达99.9%(Q30),准确检测到细胞Barcode和UMI信息的同时能够精确表征可变剪接位点、解析融合基因信息以及区分细胞亚型。
高通量
Kinnex单细胞全长转录组建库方案使用16x cDNA串联方式进行文库构建,大幅提升了全长转录组的测序通量,降低了建库测序的成本。
02 Kinnex单细胞全长转录组技术原理与流程
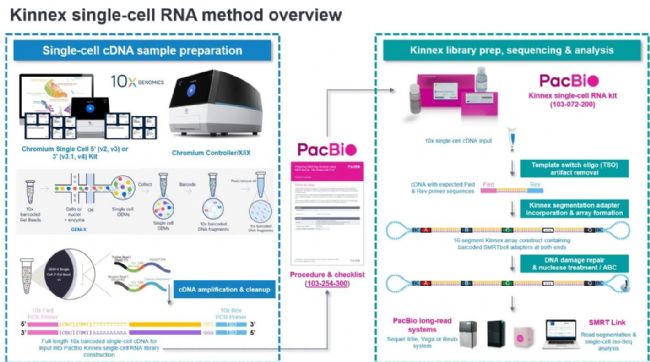
Kinnex 单细胞全长RNA试剂盒采用MAS-Seq方法来提高PacBio长读长测序的通量。MAS-Seq 是一种将cDNA分子连接成较长片段的串联方法。对串联分子测序产生的 HiFi reads 进行生物信息学拆分,即可检索到原始cDNA序列。Kinnex单细胞全长转录组建库流程以单细胞cDNA作为起始样本,支持从10x Chromium Single Cell 3' Kit(v3.1, v4)和Single Cell 5' Kit(v2, v3)生成的cDNA,作为Kinnex single-cell RNA试剂盒的输入样本,以16x cDNA串联方式连接形成有序阵列,并在两侧添加接头进行文库构建,从而提高通量,减少测序成本,实现经济高效的单细胞全长转录组测序。此外,Kinnex单细胞全长转录组建库过程中也可加入文库Barcode标签信息,支持多样本混样测序,增加了不同数据量实验需求的灵活性。
03 Kinnex single-cell RNA数据表现
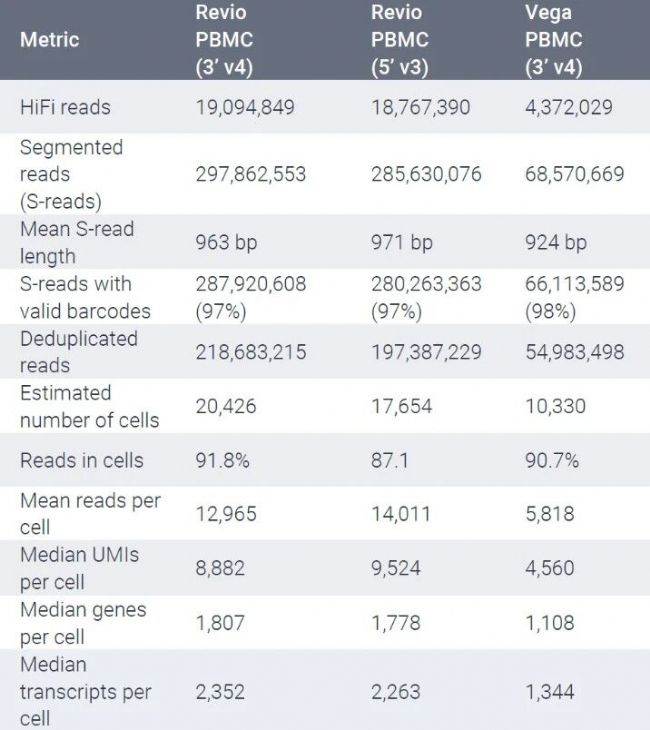
上表为使用10x Chromium GEM-X Single Cell 5' 和 3' 试剂盒生成的PBMC cDNA通过Kinnex single-cell RNA试剂盒建成的单细胞文库在PacBio的Revio和Vega长读长平台测序并使用SMRT Link中的Read Segmentation和Single-Cell Iso-Seq工作流程进行分析后得到的测试数据,结果表明,Kinnex数据具有高效的数据产出和良好的稳定性。通常情况下,在Vega和Revio系统上的单张SMRT Cell产出的HiFi reads经生信拆分还原后预计分别获得50-60M和100-120M reads的转录本数据产出。
左图为进一步使用三级生信工具CellTypist处理Revio测序得到的PBMC 10x 5' 单细胞SMRT Link输出结果的细胞聚类UMAP图,鉴定了PBMC数据集中含有的不同细胞类型。右图为同时构建HG002样本的10x 5' 单细胞Kinnex和Illumina文库后测序结果比对的Pearson相关性展示图,Pearson相关性系数为0.992,由此可见两者细胞UMI计数的高度一致性。
04 Kinnex single-cell RNA应用案例解析
案例1
题目:Detection of isoforms and genomic alterations by high-throughput full-length single-cell RNA sequencing in ovarian cancer
研究领域:肿瘤学(卵巢癌)
主要结果:
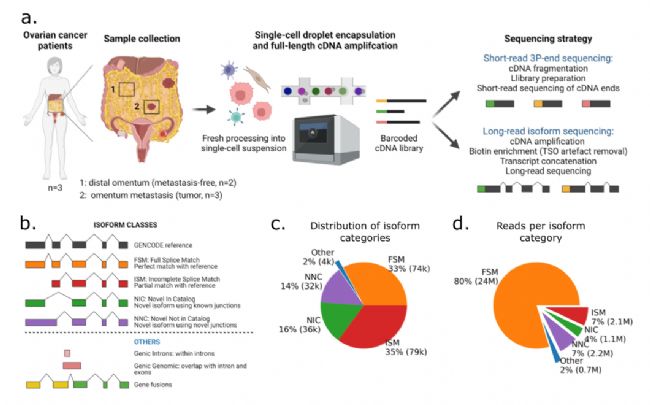
图1 研究设计概览和长读长测序数据转录异构体分析
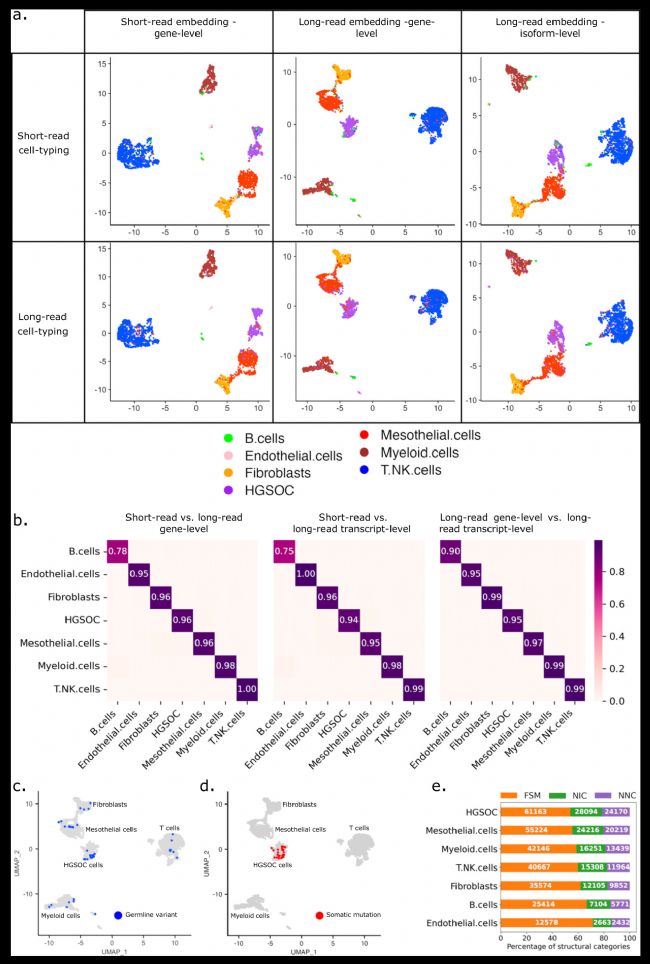
图2 细胞注释聚类和细胞类型特异性转录本异构体分布
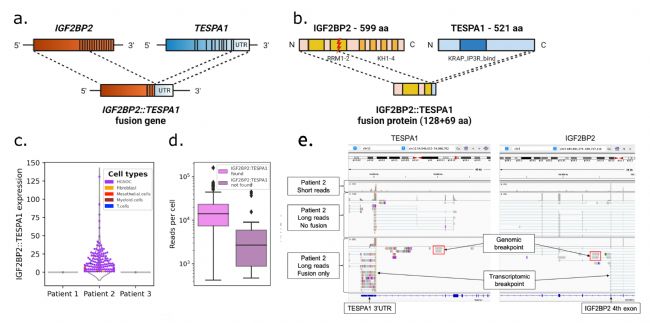
图3 肿瘤和患者特异性融合基因
IGF2BP2::TESPA1 检测
了解癌症的肿瘤内异质性及其与肿瘤微环境(TME)的复杂相互作用需要单细胞分辨率的基因型和表型信息。在瑞士苏黎世联邦理工学院研究团队的一项研究中,研究者采用10x Genomics平台进行单细胞捕获,结合串联的实验策略对网膜转移的卵巢癌患者的肿瘤和癌旁配对样本进行了基于PacBio全长的单细胞转录组测序,共检测到152,000个转录异构体,其中超过52,000个是新发现的转录异构体。该研究中,研究人员同时进行了长读长和短读长测序,发现两者单独鉴定的细胞类型和种类占比都非常相似,并表明通过PacBio的全长单细胞转录组测序可检测到细胞类型特异性转录异构体,揭示了肿瘤和间皮细胞中的差异异构体表达,还发现间皮细胞在转移中部分通过TGF-β/miR-29/胶原轴转变为癌症相关成纤维细胞。此外,研究人员通过单细胞全长转录组准确识别了融合基因,包括一种新的经过scDNA测序验证的IGF2BP2::TESPA1融合,该融合在匹配的二代短读长测序中不仅没有发现,且被错误归类为TESPA1基因高表达。这是由于二代测序读长较短,无法跨越和覆盖基因的融合位点,仅捕获到了该融合基因的 3' 端融合部分,即 TESPA1 基因部分,从而导致错误的分析结果。
案例2
题目:Developmental isoform diversity in the human neocortex informs neuropsychiatric risk mechanisms
研究领域:神经发育与疾病
主要结果:
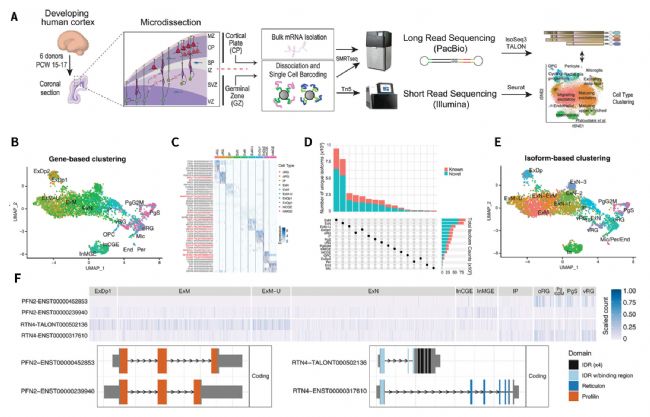
图1 妊娠中期发育中人类大脑新皮层的细胞类型特异性的全长转录组实验设计,转录异构体多样性的表征与细胞聚类和差异表达异构体分析
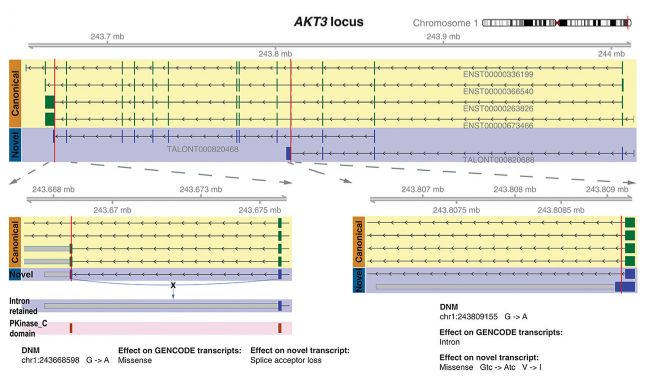
图2 以异构体为中心的神经遗传风险机制解析,
AKT3转录异构体受DNM影响的表征
人类大脑的发育受精确的分子机制调控,这些分子机制驱动时空和细胞类型特异性转录本表达程序。可变剪接是增加转录本多样性的主要机制,在人脑中非常普遍,影响大脑发育的许多方面,并与神经疾病密切相关。在加州大学一项关于神经发育的研究中,研究人员使用基于PacBio的全长转录组测序,以组织和单细胞分辨率表征了发育中的人类大脑新皮层的生发区(GZ)和皮质板(CP)区域的转录异构体多样性和神经风险机制。为了获得单细胞分辨率的转录异构体表达信息,作者使用显微切割和高保真长读长测序分析了3名受试者的受孕后第15至16周(PCW)人类大脑新皮层的GZ和CP区域的超过7000个单细胞,生成了> 26.4M的高质量PacBio CCS reads,每个细胞中平均检测到530个独特的转录本,总共比对到18,541个基因和138,497个转录异构体,其中新异构体数目占到71.7%,并将相同barcode的单细胞全长转录组数据和短读长数据结合进行聚类鉴定了发育中的人类新皮层中16个不同的细胞簇,构建了细胞类型特异性的转录异构体表达谱。通过比较不同细胞类型的转录异构体表达多样性,研究人员观察到兴奋性神经元簇,特别是那些与新生迁移神经元(ExN)和成熟神经元(ExM)相对应的神经元,拥有最多的转录异构体,突出了这些转录本在早期神经元成熟过程中的作用。通过观察在祖细胞和神经元之间动态表达的多异构体基因,发现参与肌动
蛋白聚合动力学和形态发生的
PFN2的转录异构体在祖细胞和神经元之间具有相反的表达模式,表征了不同细胞类型的异构体表达的变化和发育相关转变,以及对其编码
蛋白产物结构或稳定性的潜在作用。
此外,基于以转录异构体为核心的转录组新注释信息,研究人员对神经发育障碍(NDD)相关潜在剪接变体进行了预测,其中,来自智力残疾/发育障碍(ID/DD)的新生突变(DNM)预测会改变
AKT3新异构体(TALONT 000820468)的剪接。这种变异导致邻近剪接受体的丢失和最后一个内含子的保留,保留的内含子导致编码序列截短并丢失部分
蛋白激酶C-末端结构域。
AKT3是神经系统中PI3K-AKT-mTOR通路的关键调节剂,其失调与NDD相关。研究结果表明,多种DNM可能通过影响特定的
AKT3转录异构体而导致NDD,进一步说明一个更完整的妊娠中期大脑全长转录异构体表达目录提供了对NDD潜在遗传风险机制的更精细的分子洞察。
订购信息
PacBio Kinnex 单细胞全长转录组建库流程需要使用Kinnex single-cell RNA kit (103-072-200),前期单细胞cDNA样本制备支持搭配10x Genomics的Chromium Single Cell 3' GEM Kit(v3.1, v4)和Single Cell 5' GEM Kit(v2, v3)使用。
近年来,随着单细胞测序的深入研究,基于PacBio的单细胞全长转录组测序已在各大顶刊中崭露头角,以其在可变剪接、基因融合和突变检测的覆盖范围及测序准确性方面的巨大优势,为精准肿瘤学、癌症疫苗药物预测和神经发育与疾病等领域的复杂生物学问题解析带来了新的进展,Kinnex方法带来的高通量提升,也将使单细胞技术领域的发展推向新的高度。